Avance revolucionario en el estudio de genes bacterianos
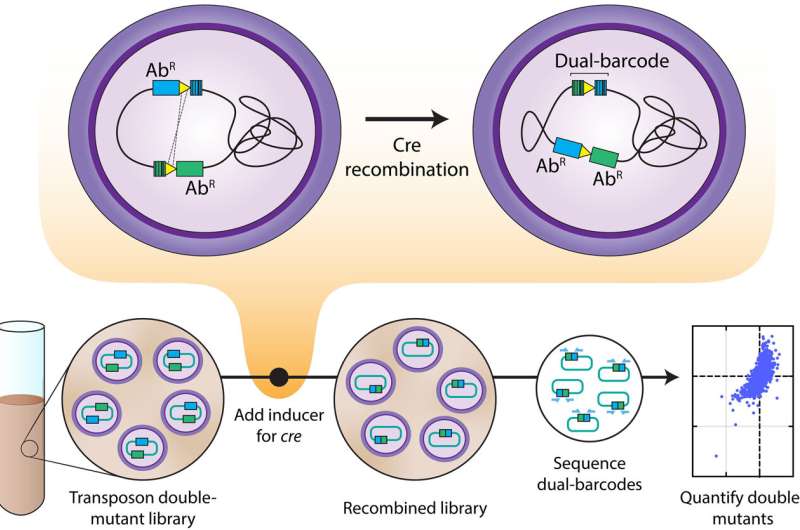
Una innovadora técnica llamada Dual Tn-seq permite identificar rápidamente las interacciones genéticas en bacterias, una clave fundamental para el desarrollo de futuros antibióticos. Investigadores de la Universidad Nacional de Singapur (NUS) y la Universidad de California, Berkeley, han descubierto cómo genes bacterianos trabajan en conjunto y detectaron vulnerabilidades críticas para atacar infecciones resistentes a medicamentos.
Cómo funciona Dual Tn-seq y sus aplicaciones
Esta metodología emplea transposones, elementos genéticos móviles marcados con códigos de barras únicos de ADN, para generar mutantes bacterianos con múltiples genes desactivados simultáneamente. Al aplicar una enzima llamada Cre recombinasa que une estas mutaciones dobles, se pueden mapear a gran escala combinaciones letales o esenciales para la bacteria, como se demostró en Streptococcus pneumoniae.
Hallazgos clave y perspectivas para nuevos medicamentos
- Identificación de 244 asociaciones génicas críticas que incluyen pares esenciales para la supervivencia bacteriana.
- Descubrimiento de la enzima PyrJ, implicada en la producción de los componentes del ADN, presente en bacterias patógenas importantes.
- Caracterización de la proteína YjbK, que actúa como un interruptor para la síntesis de la pared celular bacteriana, una estructura vital para el microorganismo.
- Asignación de posibles funciones a 67 proteínas bacterianas desconocidas mediante sus interacciones genéticas.
La técnica destacada no solo resuelve enigmas fundamentales de la genética bacteriana, sino que también tiene un gran potencial para acelerar el diseño de antibióticos dirigidos contra puntos débiles específicos, ofreciendo una esperanzadora herramienta frente a la crisis global de resistencia antimicrobiana.
Ventajas y futuro de la investigación
Dual Tn-seq es adaptable a diversas bacterias y condiciones experimentales, y no requiere colecciones mutantes previas. Además, los datos generados permitirán el entrenamiento de modelos de aprendizaje automático e inteligencia artificial para impulsar la genómica funcional en enfermedades infecciosas y biomedicina.
Como menciona el profesor Adam Deutschbauer de UC Berkeley, «Al analizar pares de genes en lugar de uno solo podemos descubrir vulnerabilidades ocultas que antes pasaban desapercibidas. Esto profundiza nuestra comprensión bacteriana y abre nuevas estrategias para combatir infecciones resistentes».
Para más información consulte el artículo original publicado en Science.
Este avance complementa otras investigaciones relacionadas en la lucha contra bacterias resistentes, como la descubierta proteína que regula el hambre y ofrece esperanza contra la obesidad, que muestran cómo la biología molecular está transformando la medicina.












Deja una respuesta